Insertionsmutagenese ist ein wesentliches Werkzeug, um die Genfunktionsanalyse im genomweiten Maßstab zu erleichtern.
Z.B. in Mais, dem Organismus, auf den wir uns konzentrieren, wurden bisher nur wenige hundert der vorhergesagten ~44.000 Mais-Genmodelle funktionell charakterisiert. Um diese Wissenslücke zu schließen, sind sequenzindizierte Reverse-Genetics-Sammlungen von Loss-of-Function-Mutanten für praktisch alle Gene jedes Genoms erforderlich.
Während für viele Arten und Insertionsstrategien Protokolle für die Mutagenese und die Generierung von flankierenden Sequenz-Tags verfügbar sind, sind keine Werkzeuge für die routinemäßige Kartierung von flankierenden Sequenzen auf Genompositionen zugänglich, die die Genannotation mit den entsprechenden Saatgutbeständen verknüpft.
Um den europäischen Forschern Loss-of-Function-Mutanten für die meisten Maisgene leichter zugänglich zu machen, war unsere Gruppe kürzlich Teil einer Anstrengung, die eine neuartige, in Europa ansässige Transposon-Insertionsbibliothek für Mutatoren (Mu) einführte, die in Plant Physiology (DOI: https://doi.org/10.1104/pp.20.00478).
Als Teil dieser Bemühungen und um die Identifizierung, Annotation und Analyse von Mu-Insertionen zu beschleunigen, haben wir eine Bioinformatik-Software namens MuWU geschrieben, die seitdem erweitert wurde, um als allgemeines Werkzeug für die nachgelagerte Analyse und Annotation der gezielten Sequenzierung von Transposon-Insertionsstellen verwendet zu werden.
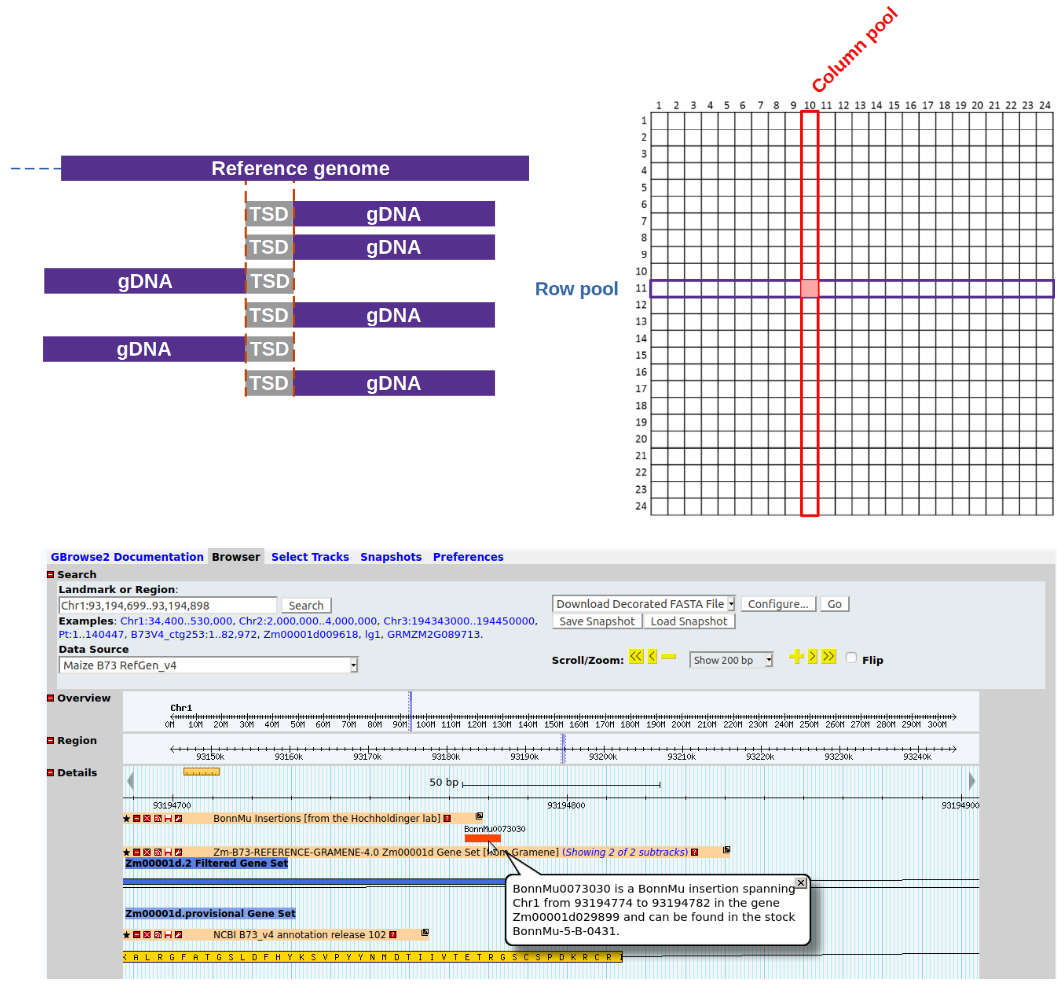
MuWU ist eine schnelle Pipeline zur Verarbeitung von Sequenzier-Reads aus einer Insertionsmutagenese wie Mu-seq. Es ermöglicht die zuverlässige Identifizierung markierter Gene und kann zwischen vererbbaren und nicht vererbbaren (somatischen) Insertionen/Mutationen unterscheiden. MuWU ordnet ferner Insertionen den entsprechenden mutierten Saatgutbeständen zu, die für Forscher auf der ganzen Welt zugänglich sind.
Unser Workflow bietet die erforderliche Robustheit und Reproduzierbarkeit, die für Routineanwendungen erforderlich sind, und führt alle Aufgaben automatisch aus, von der Verarbeitung der Rohdaten der Sequenzierung bis zur Erstellung endgültiger Ausgabetabellen. Es ist an andere Einführstrategien anpassbar. Unseres Wissens nach ist MuWU das erste offen verfügbare Tool für die Analyse von Insertionsbibliotheken, das ein multidimensionales Pooling-Design und ein gegebenes TE-Motiv nutzt, um germinale von somatischen Insertionen zu unterscheiden.
Wir glauben, dass unser Mu-seq-Workflow-Dienstprogramm von breiterem Nutzen für Forschende in der funktionellen Genetik und Bioinformatik sein wird. Einerseits für die Analyse von Insertionsbibliotheken, andererseits als Beispiel für eine neue Generation von maßgeschneiderten, Hochdurchsatzssequenz-Datenanalysen mit einem Workflow-Ansatz, der skalierbar, robust, reproduzierbar und anpassungsfähig ist.
Sowohl die Software selbst als auch die entsprechende Veröffentlichung in Oxford Bioinformatics sind unter einer Open-Source-Lizenz veröffentlicht und frei verfügbar.